はじめに
Google Researchが2025年10月16日、がん細胞の遺伝子変異を高精度で検出するAIツール「DeepSomatic」を発表しました。この技術は、腫瘍組織の遺伝子配列から、がんの進行を引き起こす変異を特定するもので、Nature Biotechnology誌に掲載された論文とともに、ツール本体とトレーニングデータセットがオープンソースとして公開されています。本稿では、この発表内容をもとに、DeepSomaticの仕組みと性能、実用上の可能性について解説します。
参考記事
メイン記事:
- タイトル: Using AI to identify genetic variants in tumors with DeepSomatic
- 著者: Kishwar Shafin (Technical Lead), Andrew Carroll (Product Lead)
- 発行元: Google Research Blog
- 発行日: 2025年10月16日
- URL: https://research.google/blog/using-ai-to-identify-genetic-variants-in-tumors-with-deepsomatic/
関連情報:
- タイトル: DeepSomatic GitHub Repository
- 発行元: Google (GitHub)
- URL: https://github.com/google/deepsomatic
- タイトル: CASTLE: Cancer Standards Long-read Evaluation
- 発行元: CASTLE Panel (GitHub)
- URL: https://github.com/CASTLE-Panel/castle
- タイトル: DeepSomatic: Accurate somatic small variant discovery for multiple sequencing technologies
- 発行元: Nature Biotechnology
- 発行日: 2025年
- URL: https://www.nature.com/articles/s41587-025-02839-x
・本稿中の画像に関しては特に明示がない場合、引用元記事より引用しております。
・記載されている情報は、投稿日までに確認された内容となります。正確な情報に関しては、各種公式HPを参照するようお願い致します。
・内容に関してはあくまで執筆者の認識であり、誤っている場合があります。引用元記事を確認するようお願い致します。
要点
- DeepSomaticは、畳み込みニューラルネットワークを用いて腫瘍細胞の体細胞変異を検出するAIツールで、Illumina、PacBio、Oxford Nanopore Technologiesの3大シーケンシングプラットフォームすべてに対応している
- 複数のベンチマークで既存手法を上回る性能を記録し、特に挿入・欠失変異の検出では、IlluminaデータでF1スコア90%、PacBioデータで80%以上を達成した
- UC Santa Cruz Genomics Instituteなどと共同で作成した高品質トレーニングデータセット「CASTLE」には、6種類のがん細胞株の全ゲノム配列データが含まれ、オープンソースとして公開されている
- 腫瘍-正常細胞ペアだけでなく、腫瘍のみのサンプルからも変異を検出可能で、小児白血病などの血液がんにも適用できる
- ツール本体、トレーニングデータセット、技術文書がすべてオープンソースで公開され、研究コミュニティが利用可能である
詳細解説
がん研究における体細胞変異の重要性
GoogleResearchによれば、がんは本質的に遺伝子疾患であり、細胞分裂の遺伝的制御が機能不全を起こすことで発生します。現在の臨床現場では、生検で採取した腫瘍細胞のゲノム配列を解析し、特定の変異に基づいて治療計画を立てる精密医療が実施されています。
がんを引き起こす遺伝子変異には、両親から受け継ぐ「生殖細胞系列変異」と、生後に獲得する「体細胞変異」の2種類があります。体細胞変異は、紫外線や化学発がん物質などの環境要因や、DNA複製時のランダムエラーによって生じます。これらの変異が細胞の正常な挙動を変化させ、本来は分裂すべきでないタイミングで細胞が増殖を始めることで、がんの初期発生や悪性化への進行を引き起こすと説明されています。
従来、Google Researchは2017年に「DeepVariant」を発表し、生殖細胞系列変異の検出で高い精度を達成しました。しかし、体細胞変異の検出はより複雑な課題です。腫瘍細胞には多様な変異が異なる頻度で含まれており、シーケンシングのエラー率が、サンプル中の体細胞変異の出現率を上回ることもあります。この技術的な困難さが、正確な変異検出の障壁となっていました。
DeepSomaticの技術的アプローチ
DeepSomaticはDeepVariantの拡張版として開発され、腫瘍細胞と正常細胞のアライメントデータ(BAMまたはCRAM形式)を入力として受け取ります。DeepVariantは生殖細胞系列変異の検出に特化していたのに対し、DeepSomaticは体細胞変異の検出に最適化されています。
DeepSomaticの処理フローは以下の通りです。まず、遺伝子配列データを画像形式に変換します。この画像には、配列データ、染色体上のアライメント、出力品質、その他の変数が含まれます。次に、畳み込みニューラルネットワークが腫瘍細胞と非がん細胞のデータを分析し、参照ゲノム、個人の生殖細胞系列変異、腫瘍の体細胞変異を区別しながら、シーケンシング過程で生じた小さなエラーを除外します。最終的に、がん関連の変異リストが標準的なVCFまたはgVCF形式で出力されます。
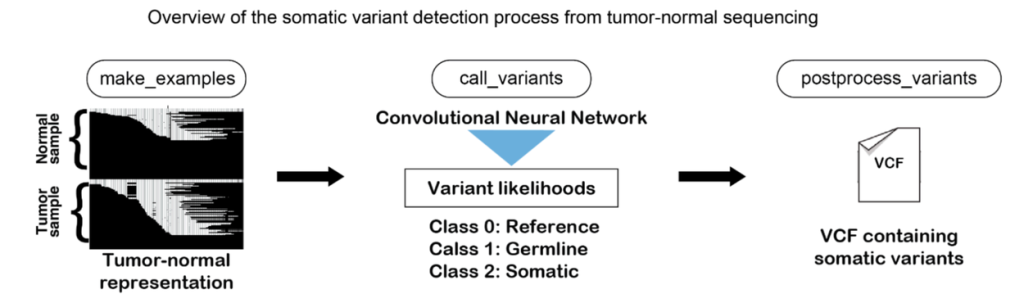
畳み込みニューラルネットワークは、画像認識で広く使われる深層学習の手法です。この技術により、DeepSomaticは配列データの視覚的なパターンから、真の変異とシーケンシングエラーを区別できると考えられます。
高品質トレーニングデータセット「CASTLE」の構築
正確なモデルの訓練には、包括的で高品質なデータと真値セットが必要です。Google Researchは、UC Santa CruzやNational Cancer Instituteなどのパートナーと共同で、4つの乳がんサンプルと2つの肺がんサンプルの腫瘍細胞と正常細胞を研究用細胞株から配列決定しました。
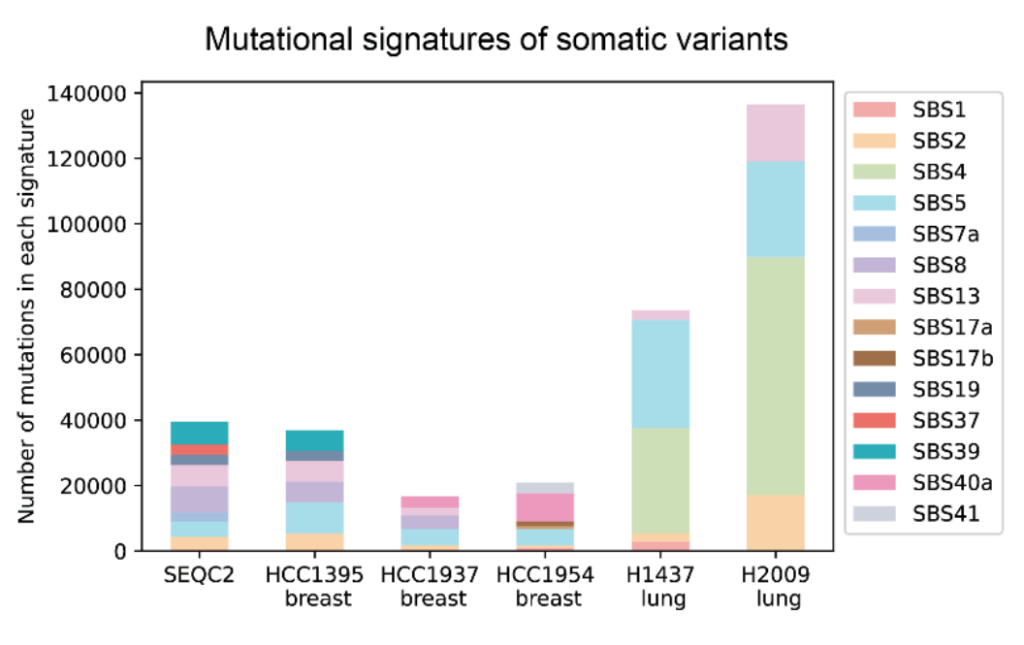
CASTLE(Cancer Standards Long-read Evaluation)データセットの特徴は、同一のサンプルを3つの主要なシーケンシングプラットフォームで解析している点です。Illuminaの短鎖読み取りシーケンシング、PacBioの長鎖読み取りシーケンシング、Oxford Nanopore Technologyの長鎖読み取りシーケンシングのデータを統合することで、プラットフォーム固有のエラーを除去し、単一の正確な参照データセットを作成しました。
CASTLEデータセットには、HCC1954、HCC1937、H1437、H2009、Hs578T、HCC1395の6つの腫瘍-正常細胞ペアが含まれています。データには、全ゲノム配列だけでなく、メチル化情報やPoreC(染色体レベルの3D構造データ)も含まれており、総データサイズは数百ギガバイトに及びます。このデータセットは、NCBI SRA BioProject(PRJNA1086849)とGoogle Cloudミラーを通じて公開されており、研究コミュニティが自由にアクセスできます。
このように複数の技術プラットフォームからのデータを統合する手法は、各プラットフォームの長所を活かしながら短所を補完できるため、より信頼性の高い参照データセットの構築に有効と考えられます。
ベンチマークでの性能評価
DeepSomaticは3つの乳がんゲノムと2つの肺がんゲノムで訓練され、訓練に含まれていない1つの乳がんゲノムと、各サンプルの1番染色体(これも訓練から除外)でテストされました。
結果として、DeepSomaticは3つの主要シーケンシングプラットフォームすべてで、既存手法を上回る性能を示しました。短鎖読み取りデータでは、SomaticSniper、MuTect2、Strelka2と比較され(SomaticSniperは一塩基変異のみ)、長鎖読み取りデータでは、合成データで訓練された深層学習モデルClairSと比較されました。
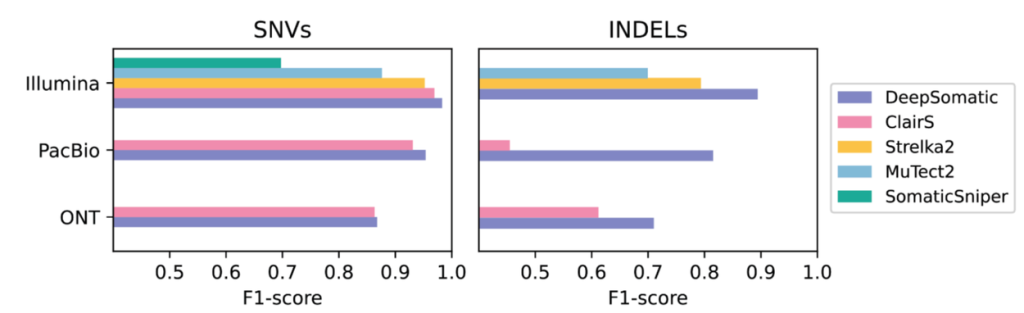
特に挿入・欠失変異(Indel)の検出で顕著な性能向上が見られました。F1スコアは、モデルが真の変異を見つける能力(再現率)と、偽陽性を出さない能力(精度)のバランスを測る指標です。Illuminaデータでは、次点の手法が80%のF1スコアだったのに対し、DeepSomaticは90%を達成しました。PacBioデータでは、次点の手法が50%未満だったのに対し、DeepSomaticは80%以上のスコアを記録しました。
一般的に、臨床応用では90%以上の精度が求められることが多いため、Illuminaデータでの90%という数値は実用レベルに近づいていると言えるでしょう。一方、PacBioデータでの80%という数値は、従来手法からは大幅な改善ですが、完全に自動化された臨床使用にはさらなる向上の余地があると考えられます。
保存サンプルとエクソーム配列への対応
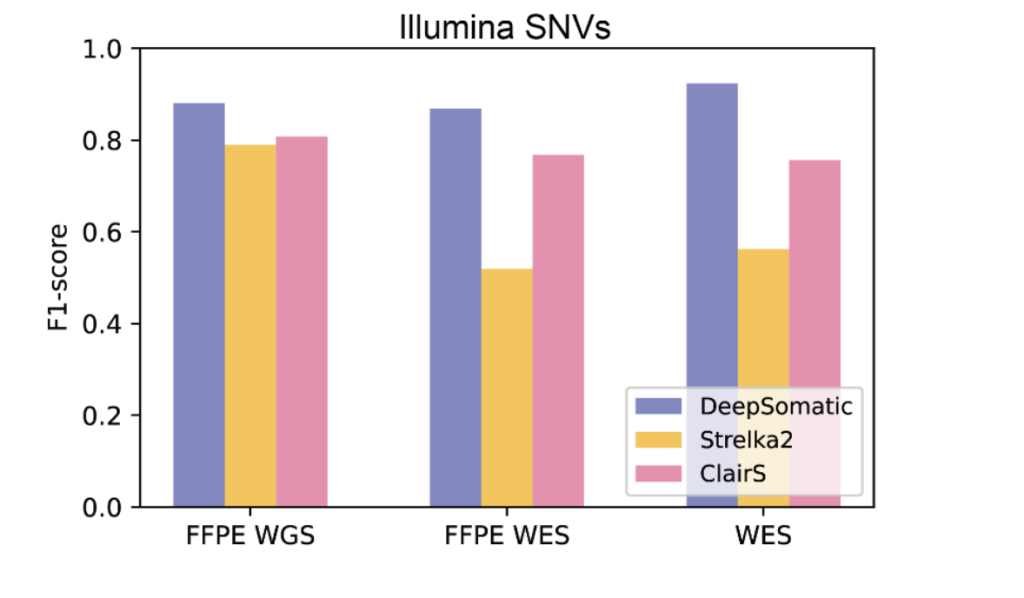
臨床や研究の現場では、ホルマリン固定パラフィン包埋(FFPE)という方法で保存された組織サンプルが広く使用されています。GoogleResearchによれば、この保存方法は追加的なDNA損傷パターンを導入するため、遺伝子解析を複雑にします。また、全ゲノム配列ではなく、タンパク質をコードするゲノムの約1%のみを対象とする全エクソーム配列(WES)も、コスト効率の良い方法として普及しています。
DeepSomaticをこれらのサンプルタイプで訓練し、訓練から除外した1番染色体でテストした結果、他のツールを上回る性能を示しました。これは、DeepSomaticが低品質または歴史的な腫瘍サンプルでも変異を特定でき、配列決定が困難だったサンプルの活用や、エクソームのみが配列決定された臨床データへの適用が可能であることを示唆しています。
FFPEサンプルへの対応は、過去に収集された膨大な保存組織を研究に活用できる可能性を開くため、重要な進展と言えるでしょう。
他のがん種への応用と腫瘍のみモード
DeepSomaticの汎用性を検証するため、訓練に含まれていない他のがん種でもテストが行われました。少数の変異から生じる攻撃的な脳腫瘍である膠芽腫の単一サンプルを解析した結果、DeepSomaticはその変異を正確に特定し、異なるがん種への学習の汎化能力を示しました。
さらに、Children’s Mercyとの共同研究では、小児白血病の8つのサンプルを解析しました。白血病は血流中に存在するため、「正常な」非がん血液サンプルを得ることができません。DeepSomaticは腫瘍のみモード(tumor-only mode)で動作でき、正常細胞の配列データがなくても体細胞変異を検出できます。この解析では、既知の変異に加えて10の新規変異が特定され、腫瘍のみサンプルでも機能することが実証されました。
腫瘍のみモードは、臨床現場で正常組織の採取が困難な場合や、過去に腫瘍サンプルのみが保存されているケースで特に有用と考えられます。
オープンソース公開と利用方法
DeepSomaticはGitHub上でオープンソースとして公開され、DeepVariantのコードベースに統合されています。主要なコンポーネントには、run_deepsomatic(実行スクリプト)、make_examples_somatic(画像データ作成)、call_variants(推論)、postprocess_variants(後処理)が含まれます。
DeepSomaticの実行には、Dockerコンテナが利用できます。基本的な使用方法は、モデルタイプ(WGS、WES、PACBIO、ONT、FFPE_WGS、FFPE_WES、WGS_TUMOR_ONLY、PACBIO_TUMOR_ONLY、ONT_TUMOR_ONLYから選択)、参照ゲノム、正常細胞と腫瘍細胞のBAMファイル、出力先を指定してrun_deepsomaticコマンドを実行する形式です。
出力はVCF形式で、FILTERフィールドに変異の種類が記載されます。「PASS」は体細胞変異、「GERMLINE」は生殖細胞系列変異、「RefCall」は参照ゲノムと同一、「LowQual」は信頼度が閾値以下、「NoCall」は深度0で判定不可を示します。この明確な分類により、研究者は体細胞変異を効率的に識別できます。
Illumina、PacBio、Oxford Nanopore Technologiesの各プラットフォーム、および全ゲノム配列・全エクソーム配列、腫瘍-正常ペア・腫瘍のみなど、様々な組み合わせでのケーススタディが詳細に記載されています。実行時間や精度の期待値についても、メトリクスページで確認できます。
Dockerコンテナの利用により、環境構築の手間を削減し、研究者が迅速にツールを試せる点は、実用化を促進する上で重要と考えられます。
今後の展開と期待
研究室や臨床医がこのツールを使い始めることへの期待がGoogleResearchからは表明されています。既知のがん変異の検出は、化学療法、免疫療法、その他の方法から既存治療を選択する際に役立つ可能性があります。新規のがん変異の特定は、全く新しい治療法の開発につながるかもしれません。
DeepSomaticはBSD-3-Clauseライセンスで公開されており、研究・商用の両方で利用可能です。ただし、免責事項として、このツールは医療機器として意図されておらず、診断や予後判定を含むいかなる臨床使用も意図されていないことが明記されています。現時点では研究用途に限定されていますが、将来的には臨床グレードのツールへの発展も期待できるでしょう。
Google Researchは、マンモグラムによる乳がん検診、CTスキャンによる肺がん検診、婦人科がんの研究推進など、がん関連のAI研究を幅広く展開しています。DeepSomaticは、これらの取り組みの一環として、がん研究の加速と精密医療の実現を目指すものです。
まとめ
DeepSomaticは、畳み込みニューラルネットワークを用いて腫瘍細胞の体細胞変異を高精度で検出するAIツールです。3大シーケンシングプラットフォームすべてに対応し、既存手法を上回る性能を示しました。特に重要なのは、ツール本体と高品質なトレーニングデータセットの両方がオープンソースで公開され、研究コミュニティが自由に利用・改良できる点です。臨床応用にはさらなる検証が必要ですが、がん研究の加速と精密医療の実現に向けた重要な一歩と言えるでしょう。今後、このツールがどのように発展し、実際の医療現場で活用されていくのか、注目していきたいところです。









