はじめに
近年、生命科学分野では、個々の細胞が持つ遺伝子発現情報を網羅的に解析するシングルセルRNAシーケンシング(scRNA-seq)技術が注目されています。これにより、細胞一つひとつの個性や機能を詳細に理解することが可能になりました。しかし、scRNA-seqから得られるデータは膨大かつ高次元であり、その解析には専門的な知識とツールが必要で、時間と労力がかかるという課題がありました。
本稿では、Google ResearchとYale Universityが発表した、この課題に対する画期的なアプローチであるCell2Sentence-Scale(C2S-Scale)を紹介します。C2S-Scaleは、大規模言語モデル(LLM)を活用し、シングルセルデータを「言語」として扱うことで、解析の効率化、スケーラビリティの向上、そして専門家以外でも扱いやすくすることを目指したモデルファミリーです。技術的な側面を中心に、背景となる前提知識も交えながら詳細に解説します。
引用元情報
- 記事タイトル: Teaching machines the language of biology: Scaling large language models for next-generation single-cell analysis
- 発行日: 2025年4月17日
- 参照元URL: https://research.google/blog/teaching-machines-the-language-of-biology-scaling-large-language-models-for-next-generation-single-cell-analysis/
要点
- 課題: シングルセルデータ(scRNA-seq)は膨大・高次元で解釈が難しく、解析に専門知識が必要。
- 提案: C2S-Scaleは、シングルセルデータを「細胞文(Cell Sentence)」というテキスト形式に変換し、LLMで解析する手法。
- 基盤モデル: Googleのオープンモデル「Gemma」をベースに、生物学データと言語による推論能力を統合。
- モデルファミリー: 計算リソースやタスクに応じて選択可能な、4億から270億パラメータまでの複数モデルを提供(オープンソース)。
- 主な機能:
- 対話型解析: 自然言語での質問応答(例:「このT細胞は抗PD-1療法にどう反応するか?」)。
- データ解釈: 細胞や組織に関する生物学的要約の自動生成。
- 応答予測: 薬剤投与や遺伝子ノックアウトなど、摂動(perturbation)に対する細胞応答のシミュレーション。
- スケーリング則: モデルサイズが大きいほど性能が向上するスケーリング則を確認。
- 強化学習: 生物学的な正しさを評価する報酬関数(BERTScoreなど)を用いたファインチューニング。
詳細解説
1. 前提知識:シングルセル解析の課題とLLM活用の着想
私たちの体は多様な機能を持つ細胞で構成されています。scRNA-seqは、個々の細胞内でどの遺伝子がどれだけ活発に働いているか(遺伝子発現量)を測定する技術です。これにより、例えば同じ組織内でも細胞ごとの微妙な違いや状態を捉えることができます。
しかし、一つの細胞から得られる遺伝子発現データは数千次元にも及び、実験対象によっては数万〜数百万細胞のデータセットになります。この高次元性・大規模性が、従来の解析手法におけるボトルネックでした。データ構造が複雑なため、特徴抽出や次元削減、クラスタリングなどに専門的なバイオインフォマティクスの知識や専用ツールが不可欠だったのです。
ここで、LLMの持つ自然言語処理能力と文脈理解能力に着目したのがC2S-Scaleのアプローチです。もし、細胞の遺伝子発現プロファイルを、人間やLLMが理解できる「言語」に変換できれば、解析のハードルは劇的に下がるのではないか?という発想です。遺伝子の名称や細胞の種類、実験条件といった生物学情報の多くは元々テキストで表現されているため、LLMとの親和性は高いと考えられます。
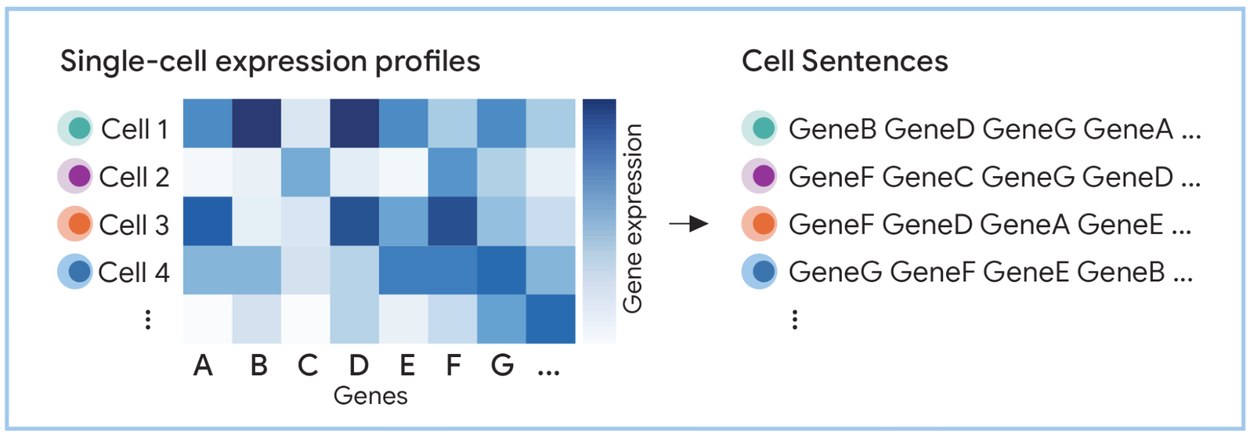
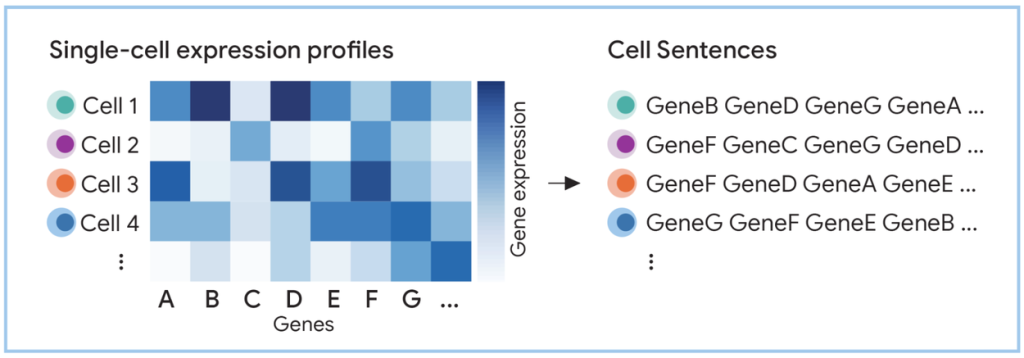
2. 細胞から文へ:Cell Sentenceの生成
C2S-Scaleの中核となるアイデアが「細胞文(Cell Sentence)」です。これは、個々の細胞の遺伝子発現プロファイルをテキストシーケンスに変換する手法です。具体的には、各細胞において発現レベルが高い遺伝子を順番に並べたリストを生成します。
例えば、ある細胞でGeneB, GeneD, GeneG, GeneA… の順に発現量が高かった場合、その細胞のCell Sentenceは “GeneB GeneD GeneG GeneA …” となります。これにより、数千次元の数値ベクトルであった細胞データが、LLMが直接処理可能なテキストデータに変換されます。
このテキスト化により、以下のような利点が生まれます。
- アクセシビリティ向上: 自然言語インターフェースを通じて、専門家以外でもデータにアクセスしやすくなります。
- 解釈性向上: LLMによる要約や説明生成が可能になり、データの意味するところを理解しやすくなります。
- 柔軟性向上: テキスト化された生物学的情報(遺伝子名、細胞種、文献情報など)と統合しやすくなります。
3. C2S-Scaleモデルアーキテクチャと学習
C2S-Scaleは、GoogleのオープンモデルファミリーであるGemmaを基盤としています。Gemmaの強力な言語理解能力を維持しつつ、生物学的な推論能力を獲得させるために、Cell Sentence、メタデータ、関連する生物学的文脈を統合したプロンプト設計と、大規模なデータセットによる学習が行われました。
- 学習データ: 実際のscRNA-seqデータセットから生成されたCell Sentence、生物学的メタデータ、科学文献など、10億トークンを超えるデータで学習されています。
- モデルサイズ: 研究コミュニティの多様なニーズに応えるため、410M(4億1千万)から27B(270億)パラメータまでの複数のモデルが用意されています。小規模モデルは計算効率が高く、限られたリソースでのファインチューニングやデプロイに適しています。大規模モデルは計算コストが高いものの、より高い性能を発揮します。これらのモデルはオープンソースとして公開され、研究者が自由に利用・改変できます。
- アーキテクチャ: 基盤となるLLMアーキテクチャは変更されておらず、既存のLLMインフラやエコシステムの恩恵を最大限に活用できます。
4. C2S-Scaleの能力と応用
- 対話型解析(Question Answering):
ユーザーが「このT細胞は抗PD-1療法(がん治療薬)にどう反応しますか?」といった自然言語の質問を投げかけると、C2S-ScaleはCell Sentenceデータと事前学習で得た生物学的知識を統合して回答を生成します。これにより、コードを書かずに対話形式でデータ探索を進めることが可能になります。論文の図では、他のLLMと比較して高い性能(BioBERT Score)を示しています。
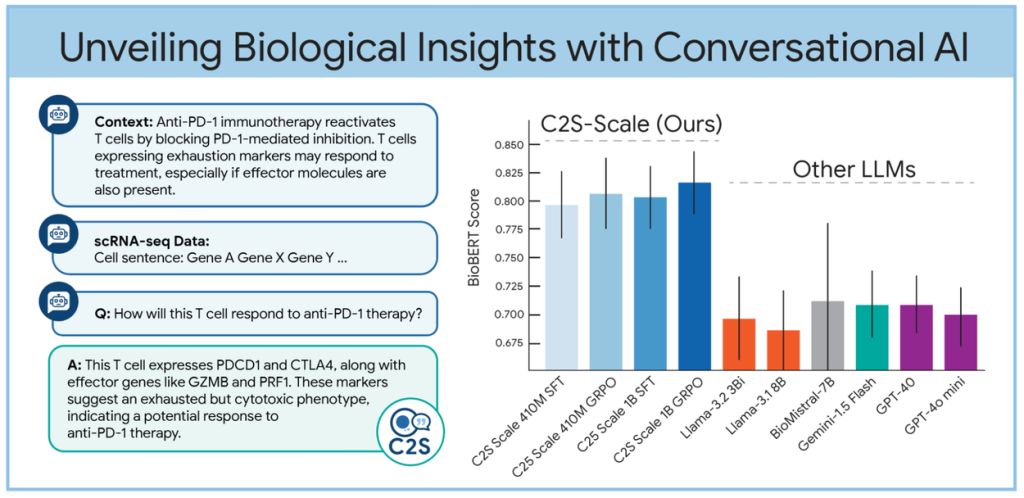
- 自然言語によるデータ解釈:
単一細胞の細胞タイプの記述から、組織全体や実験全体の生物学的要約まで、様々な粒度でscRNA-seqデータのサマリーを自動生成します。これにより、研究者は新しいデータセットを迅速かつ自信を持って解釈できます。
- 生物学におけるスケーリング則:
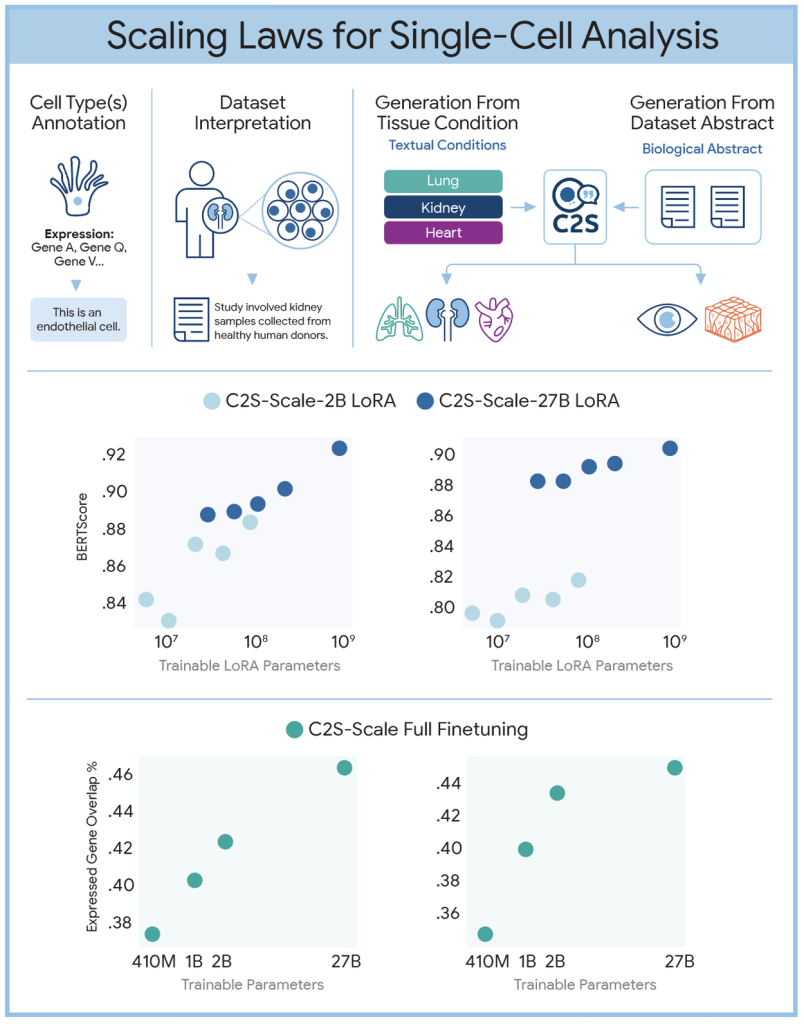
本研究の重要な発見の一つは、生物学的言語モデルにおいても明確なスケーリング則が成り立つことです。つまり、モデルサイズ(パラメータ数)を大きくするほど、細胞タイプの注釈付け、細胞や組織の生成といった様々なタスクにおいて性能が一貫して向上します。これは、汎用LLMで見られる傾向と同様であり、より多くのデータと計算資源を投入することで、生物学的LLMがさらに進化し続ける可能性を示唆しています。論文の図では、パラメータ効率の良いファインチューニング(LoRA)とフルファインチューニングの両方で、モデルサイズ増大に伴う性能向上(BERTScoreや遺伝子オーバーラップ率の改善)が示されています。
- 細胞応答予測(Perturbation Prediction):
C2S-Scaleの特に有望な応用が、摂動(薬剤投与、遺伝子ノックアウト、サイトカイン曝露など)に対する細胞の応答予測です。ベースラインとなる細胞のCell Sentenceと処置内容を入力として与えると、モデルは応答後の遺伝子発現変化を反映した新しいCell Sentenceを生成します。
このようなin silico(計算機上)での細胞挙動シミュレーションは、創薬、個別化医療、実験優先順位付けなどを加速させる可能性があります。これは、次世代のモデルシステムとして提案されている「仮想細胞(Virtual Cells)」の実現に向けた大きな一歩であり、従来の細胞株や動物モデルに代わる、より高速、安価、かつ倫理的な代替手段となる可能性を秘めています。
- 強化学習による最適化:
GeminiのようなLLMが人間の指示に従い、有用な応答を生成するために強化学習でファインチューニングされるのと同様に、C2S-Scaleも生物学的推論に最適化するために同様の技術が適用されています。生物学的なテキスト評価に適した報酬関数(例:BERTScore)を用いることで、データセット内の実際の応答に近い、生物学的に正確で有益な出力を生成するようにモデルを訓練します。これにより、特に治療介入モデリングのような複雑なタスクにおいて、科学的発見に役立つ応答へとモデルを誘導します。
まとめ
本稿では、シングルセルデータを「言語」として扱い、LLMの能力を最大限に活用する新しいアプローチ、C2S-Scaleを紹介しました。Cell Sentenceという革新的なデータ表現、Gemmaベースの強力なモデルアーキテクチャ、そしてスケーリング則の確認により、C2S-Scaleはシングルセル解析の可能性を大きく広げます。 対話型解析、自動要約、応答予測といった機能は、生物学研究の効率化と民主化を促進し、創薬や個別化医療への貢献も期待されます。モデルとリソースはオープンソースとして公開されており、今後のコミュニティによる活用と発展が楽しみです。C2S-Scaleは、機械に生命の言語を教えることで、生物学の未解決問題に挑むための強力なツールとなるでしょう。